Halicin
Halicin (SU-3327) ist eine heterocyclische Verbindung und ein Enzymhemmer gegen c-Jun-N-terminale Kinasen (JNK).[2]
| Strukturformel | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
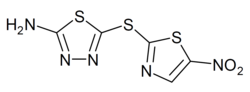
| ||||||||||||||||
| Allgemeines | ||||||||||||||||
| Name | Halicin | |||||||||||||||
| Andere Namen |
| |||||||||||||||
| Summenformel | C5H3N5O2S3 | |||||||||||||||
| Externe Identifikatoren/Datenbanken | ||||||||||||||||
| ||||||||||||||||
| Eigenschaften | ||||||||||||||||
| Molare Masse | 261,3 g·mol−1 | |||||||||||||||
| Sicherheitshinweise | ||||||||||||||||
| ||||||||||||||||
| Soweit möglich und gebräuchlich, werden SI-Einheiten verwendet. Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen (0 °C, 1000 hPa). | ||||||||||||||||
Ursprünglich für die Behandlung von Diabetes erwogen, kam Halicin als ein solches Medikament jedoch nie auf den Markt.[3][4]
Im Jahr 2019 wurde Halicin jedoch von Forschern am Massachusetts Institute of Technology unter Verwendung eines In-Silico-Deep-Learning-(KI)-Ansatzes als potenzielles Breitbandantibiotikum identifiziert.[5][3][6] Benannt wurde es nach HAL, dem fiktiven Computer des Films 2001: Odyssee im Weltraum.[5] Nach Aussagen von Studienautoren ist es das erste Antibiotikum, das ohne jede Prämisse durch künstliche Intelligenz entdeckt wurde.[5] Die Wirksamkeit von Halicin in lebenden Organismen wurde anschließend durch In-vitro-Zellkulturtests, gefolgt von In-vivo-Tests an Labormäusen bestätigt. Halicin wirkte in Tests gegen bislang multiresistente Bakterien wie Clostridium difficile, Acinetobacter baumannii und Mycobacterium tuberculosis mit einem bisher ungewöhnlichen Mechanismus, der vermutlich die Sequestrierung von Eisen in den Bakterienzellen beinhaltet und dadurch deren Fähigkeit beeinträchtigt, das pH-Gleichgewicht über die Zellmembran hinweg richtig zu regulieren. Anders als die meisten Antibiotika konnte Halicin seine Wirkung gegen Bakterienstämme beibehalten, die gegen alle bisherigen Medikamente resistent sind.[3]
Eine Arzneimittelzulassung ist mit Stand 2020 nicht erfolgt, präklinische Studien sind geplant.[3]
Einzelnachweise
Bearbeiten- ↑ a b Datenblatt JNK Inhibitor XIII - CAS 40045-50-9 - Calbiochem, The JNK Inhibitor XIII, also referenced under CAS 40045-50-9, controls the biological activity of JNK. This small molecule/inhibitor is primarily used for Phosphorylation & Dephosphorylation applications. bei Sigma-Aldrich, abgerufen am 23. Februar 2020 (PDF).
- ↑ Sehwan Jang, Li-Rong Yu, Mohamed A. Abdelmegeed, Yuan Gao, Atrayee Banerjee: Critical role of c-jun N-terminal protein kinase in promoting mitochondrial dysfunction and acute liver injury. In: Redox Biology. Band 6, 9. Oktober 2015, S. 552–564, doi:10.1016/j.redox.2015.09.040, PMID 26491845, PMC 4625008 (freier Volltext).
- ↑ a b c d Julia Merlot: Kampf gegen Resistenzen: Künstliche Intelligenz entdeckt vielversprechendes Antibiotikum. DER SPIEGEL, abgerufen am 22. Februar 2020.
- ↑ Surya K. De, John L. Stebbins, Li-Hsing Chen, Megan Riel-Mehan, Thomas Machleidt: Design, Synthesis, and Structure-Activity Relationship of Substrate Competitive, Selective, and in Vivo Active Triazole and Thiadiazole inhibitors of the c-Jun N-Terminal Kinase. In: Journal of Medicinal Chemistry. Band 52, Nr. 7, 9. April 2009, S. 1943–1952, doi:10.1021/jm801503n, PMID 19271755, PMC 2667321 (freier Volltext).
- ↑ a b c Jo Marchant: Powerful antibiotics discovered using AI. In: Nature. 20. Februar 2020, doi:10.1038/d41586-020-00018-3.
- ↑ Jonathan M. Stokes, Kevin Yang, Kyle Swanson, Wengong Jin, Andres Cubillos-Ruiz: A Deep Learning Approach to Antibiotic Discovery. In: Cell. Band 180, Nr. 4, 20. Februar 2020, S. 688–702.e13, doi:10.1016/j.cell.2020.01.021, PMID 32084340.