Basenpaar
Als Basenpaar bezeichnet man im Doppelstrang einer doppelsträngigen Nukleinsäure (DNA oder RNA, in diesem Fall nach englisch double stranded auch als dsDNA bzw. dsRNA bezeichnet) zwei gegenüberliegende Nukleobasen, die zueinander komplementär sind und durch Wasserstoffbrückenbindungen zusammengehalten werden.

Während die Länge einzelsträngiger Nukleinsäuren (ssRNA oder ssDNA, englisch single stranded) mit der Zahl an Nukleotiden (nt) oder Basen (b) – im synthetischen Fall gelegentlich auch mit der allgemeinen Bezeichnung -mer für die Anzahl der Sequenzen eines Kettenmoleküls[1] – gegeben ist, wird die Größe von doppelsträngigen DNA-Abschnitten meist in Basenpaaren angegeben, abgekürzt bp,
- 1 nt = ein Nukleotid (im Einzelstrang)
- 1 bp = ein Basenpaar (im Doppelstrang)
- 1 kbp oder kb (Kilo-Basenpaare) = 1000 (103) Basenpaare
- 1 Mbp oder Mb (Mega-Basenpaare) = 1.000.000 (106) Basenpaare
- 1 Gbp oder Gb (Giga-Basenpaare) = 1.000.000.000 (109) Basenpaare
Das unverdoppelte haploide menschliche Genom im Zellkern einer Keimzelle umfasst über 3 Milliarden Basenpaare, etwa 3,2 Gbp, verteilt auf 23 Chromosomen (1n; 1c). Eine somatische Zelle des menschlichen Körpers enthält gewöhnlich einen diploiden (zweifachen) nukleären Chromosomensatz, also etwa 6,4 Gbp, auf 46 Chromosomen (2n; 2c). Dieser wird vor einer Zellteilung dupliziert (verdoppelt), so dass jedes der 46 Chromosomen aus zwei Chromatiden – einander gleiche Kopien mit derselben genetischen Information – besteht, bevor die Kernteilung als Mitose beginnt, mit ungefähr 13 Gbp (2n; 4c). Neben dieser nukleären DNA (Kern-DNA, nDNA) enthalten die meisten menschlichen Zellen wie bei allen Eukaryonten in jedem Mitochondrium noch ein weiteres Genom (Mitogenom), von je etwa 16,6 kbp (mitochondriale DNA, mtDNA). Eine Ausnahme sind die reifen roten Blutkörperchen, die wie bei allen Säugetieren weder Zellkern noch Mitochondrien aufweisen. Pflanzliche Zellen enthalten über dieses hinaus noch das Plastiden-Genom (Plastom) ihrer Chloroplasten[2] (abgekürzt ctDNA[3] oder cpDNA[4]).
Die Anzahl der Basenpaare ist auch ein wichtiges Maß für den Umfang an Information, die in einem Gen gespeichert ist. Da jedes Basenpaar eine Wahl aus 4 möglichen Formen darstellt, entspricht 1 bp dem Informationsgehalt von 2 bit, dem Doppelten eines Bits im Binärcode. Allerdings trägt im menschlichen Genom nur ein geringer Teil der DNA die genetische Information für den Aufbau von Proteinen; über 95 % sind nichtcodierend und bestehen häufig aus repetitiven Elementen.
Bedeutung
BearbeitenDie Basenpaarung spielt eine wesentliche Rolle für die DNA-Reduplikation, für die Transkription und die Translation im Zuge der Proteinbiosynthese sowie für vielfältige Ausgestaltungen der Sekundärstruktur und Tertiärstruktur von Nukleinsäuren.
- Bei der Replikation wird der DNA-Doppelstrang aufgetrennt und die beiden komplementären Einzelstränge werden durch Basenpaarung aus Desoxyribonukleotiden zu zwei DNA-Doppelsträngen ergänzt.
- Bei der Transkription wird ein codogener Strangabschnitt der DNA als Vorlage genutzt, um durch Basenpaarung aus Ribonukleotiden einen RNA-Einzelstrang mit komplementärer Basensequenz aufzubauen, wobei A mit U gepaart wird. Die gebildeten RNA-Stränge dienen als mRNA, als tRNA oder als rRNA verschiedenen Aufgaben.
- Bei der Translation wird die Basensequenz eines mRNA-Abschnitts in Dreierschritten abgelesen, indem sich jeweils die drei Basen des Anticodons von tRNAs mit den komplementären Basentripletts der mRNA paaren. Die in DNA gespeicherte und in mRNA umgeschriebene Basensequenz wird mit den von tRNA transportierten Aminosäuren so in eine Sequenz von Aminosäuren übersetzt und codiert also die Aminosäuresequenz als die Primärstruktur eines Proteins. Hierbei treten auch die Wobble-Paarungen bei der Paarung der 3. Base eines Codons der mRNA mit der 1. Base der tRNA auf.
Paarungsregeln
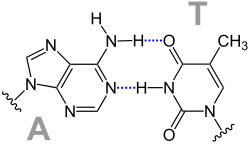
BearbeitenGebildet wird ein Basenpaar durch Wasserstoffbrückenbindung zwischen zwei Nukleobasen. Dabei wird eine der Purinbasen Guanin oder Adenin mit einer der Pyrimidinbasen Cytosin, Thymin oder Uracil zu einem Paar verbunden. Bei den komplementären Basenpaarungen zwischen zwei Strangabschnitten von Nukleinsäuren bildet Guanin mit Cytosin ein Paar sowie Adenin mit Thymin oder mit Uracil. Daraus können sich folgende Paarungen ergeben:
DNA/DNA
- Guanin mit Cytosin: G-C bzw. C-G
- Adenin mit Thymin: A-T bzw. T-A
DNA/RNA
- Guanin mit Cytosin: G-C bzw. C-G
- Adenin mit Thymin: T-A
- Adenin mit Uracil: A-U
RNA/RNA
- Guanin mit Cytosin: G-C bzw. C-G
- Adenin mit Uracil: A-U bzw. U-A
Watson-Crick-Paarungen
BearbeitenBereits 1949 stellte der österreichische Biochemiker Erwin Chargaff mit den Chargaff-Regeln fest, dass in der DNA die Anzahl der Basen Adenin (A) und Thymin (T) stets im Verhältnis 1 : 1 vorliegt, ebenso beträgt das Verhältnis der Basen Guanin (G) und Cytosin (C) 1 : 1. Dagegen variiert das Mengenverhältnis A : G beziehungsweise C : T stark.
Daraus schlossen James D. Watson und Francis Harry Compton Crick, dass A-T und G-C jeweils komplementäre Basenpaare bilden.
In der tRNA und rRNA treten ebenfalls Basenpaarungen auf, wenn der Nukleotid-Strang Schleifen bildet und sich dadurch komplementäre Basensequenzen gegenüberstehen. Da in der RNA statt Thymin nur Uracil eingebaut wird, sind die Paarungen A-U und G-C.
Ungewöhnliche Paarungen
BearbeitenUngewöhnliche Paarungen treten vor allem in tRNAs und in Tripelhelices auf. Sie folgen zwar dem Watson-Crick-Schema, bilden aber andere Wasserstoffbrückenbindungen aus: Beispiele sind Reverse-Watson-Crick-Paarungen, Hoogsteen-Paarungen (benannt nach Karst Hoogsteen, geboren 1923) und Reverse-Hoogsteen-Paarungen
Nicht-Watson-Crick-Basenpaare mit Watson-Crick-ähnlicher Geometrie
BearbeitenBereits Ende des 20. Jahrhunderts zeigten verschiedene Studien Anhaltspunkte für die Existenz von Nicht-Watson-Crick-Basenpaaren mit Watson-Crick-ähnlicher Geometrie bei der Interaktion von tRNA und mRNA, wenn diese Pseudouridin(Ψ) oder Inosin(I) enthalten.
Der tRNA-Rest befindet sich bei dieser Darstellung dabei immer an Position 34, das mRNA-Gegenstück an der Position +3. Für die A-Ψ-Bindung weichen diese Werte ab und sind entsprechend gekennzeichnet.
„■“ indiziert die Verwendung der Hoogsteen-Seite (cis), „⬤“ die der Watson-Crick-Seite (cis). Eine Vermittlung der Basenpaarung durch Wasser wird durch ein „W“ in der Paarung signalisiert. Ein „~“ zeigt die Notwendigkeit einer tautomeren Base an. „*“ kennzeichnet modifizierte Basen.[5]
| tRNA-Rest | mRNA-Rest | Art der Basenpaarung |
|---|---|---|
| Ψsyn[6] | A | ■―⬤ |
| Gsyn[7] | G | ■W⬤ |
| Gsyn[7] | A+ | ■―⬤ |
| Gsyn[7] | A+ | ■―⬤ |
| G[7] | Gsyn | ■~⬤ |
| G[7] | Gsyn | ⬤W■ |
| G[7] | Asyn | ⬤―■ |
| I | Asyn | ⬤―■ |
| I | Gsyn | ⬤~■ |
| I | Gsyn | ⬤W■ |
| Ψ[6] | A | Watson-Crick |
| U* | G | Watson-Crick (U~C) |
| C*[8][9][10] | A | Watson-Crick (C~A) |
| A (36)[11] | Ψ (+1) | Watson-Crick |
| A (36)[11] | Ψsyn (+1) | ⬤―■ |
Damit die Bindungen U⬤―■A und C⬤―■G ausgebildet werden können, muss C entweder in der imino-Form vorliegen oder protoniert sein.[5]
Wobble-Paarungen
BearbeitenDie Bezeichnung bezieht sich auf die Wobble-Hypothese von Francis Crick (1966). Wobble-Paarungen sind die Nicht-Watson-Crick-Paarungen G-U oder G-T und A-C:
Paarungen synthetischer Basen
BearbeitenIn der synthetischen Biologie werden u. a. Nukleinsäuren mit synthetische Basen erzeugt und untersucht, teilweise auch mit dem Ziel von Paarungen dieser Basen. Ein Beispiel ist die Hachimoji-DNA.
Weblinks
Bearbeiten- Eine große Datenbank mit zahlreichen Strukturen von Basenpaaren (Abteilung Biologie und Biochemie, University of Houston)
Einzelnachweise
Bearbeiten- ↑ Vgl. Dimer, Trimer, Oligomer, Polymer, siehe etwa Pegaptanib, ein 27mer.
- ↑ Hogan, C. Michael (2010). Deoxyribonucleic acid. Encyclopedia of Earth. National Council for Science and the Environment. Hrsg. S. Draggan, C. Cleveland. Washington DC
- ↑ ctDNA — chloroplast DNA. AllAcronyms.com, archiviert vom am 3. Juni 2013.
- ↑ The Oxford Dictionary of Abbreviations. ctDNA—Dictionary definition 1998 (encyclopedia.com).
- ↑ a b c Isostericity and tautomerism of base pairs in nucleic acids. In: FEBS Letters. Band 588, Nr. 15, 1. August 2014, ISSN 0014-5793, S. 2464–2469, doi:10.1016/j.febslet.2014.06.031 (sciencedirect.com [abgerufen am 28. Oktober 2020]).
- ↑ a b Bruno Senger, Sylvie Auxilien, Uwe Englisch, Friedrich Cramer, Franco Fasiolo: The Modified Wobble Base Inosine in Yeast tRNAIle Is a Positive Determinant for Aminoacylation by Isoleucyl-tRNA Synthetase. In: Biochemistry. Band 36, Nr. 27, 1. Juli 1997, ISSN 0006-2960, S. 8269–8275, doi:10.1021/bi970206l.
- ↑ a b c d e f Karin Zerfass, Hildburg Beier: Pseudouridine in the anticodon GΨA of plant cytoplasmic tRNA Tyr is required for UAG and UAA suppression in the TMV-specific context. In: Nucleic Acids Research. Band 20, Nr. 22, 25. November 1992, ISSN 0305-1048, S. 5911–5918, doi:10.1093/nar/20.22.5911, PMID 1461724, PMC 334454 (freier Volltext) – (oup.com [abgerufen am 28. Oktober 2020]).
- ↑ Debabrata Mandal, Caroline Köhrer, Dan Su, Susan P. Russell, Kady Krivos: Agmatidine, a modified cytidine in the anticodon of archaeal tRNAIle, base pairs with adenosine but not with guanosine. In: Proceedings of the National Academy of Sciences. Band 107, Nr. 7, 16. Februar 2010, ISSN 0027-8424, S. 2872–2877, doi:10.1073/pnas.0914869107, PMID 20133752, PMC 2840323 (freier Volltext) – (pnas.org [abgerufen am 28. Oktober 2020]).
- ↑ Yoshiho Ikeuchi, Satoshi Kimura, Tomoyuki Numata, Daigo Nakamura, Takashi Yokogawa: Agmatine-conjugated cytidine in a tRNA anticodon is essential for AUA decoding in archaea. In: Nature Chemical Biology. Band 6, Nr. 4, April 2010, ISSN 1552-4469, S. 277–282, doi:10.1038/nchembio.323.
- ↑ T. Muramatsu, S. Yokoyama, N. Horie, A. Matsuda, T. Ueda: A novel lysine-substituted nucleoside in the first position of the anticodon of minor isoleucine tRNA from Escherichia coli. In: The Journal of Biological Chemistry. Band 263, Nr. 19, 5. Juli 1988, ISSN 0021-9258, S. 9261–9267, doi:10.1351/pac198961030573, PMID 3132458.
- ↑ a b John Karijolich, Yi-Tao Yu: Converting nonsense codons into sense codons by targeted pseudouridylation. In: Nature. Band 474, Nr. 7351, Juni 2011, ISSN 1476-4687, S. 395–398, doi:10.1038/nature10165, PMID 21677757, PMC 3381908 (freier Volltext).