Großes T-Antigen
Das große T-Antigen (LTag oder LT, von engl. large ‚groß‘ und Tumor-Antigen) ist ein onkogenes und DNA-bindendes Protein des Simian-Virus 40 (SV40). Es wird in der Biochemie und Zellbiologie zur Immortalisierung von Säugetier-Zellen verwendet.[1][2] Homologe kommen in anderen Polyomaviren vor,[3] mit Längen zwischen 600 und 800 Aminosäuren.
| Großes T-Antigen | ||
|---|---|---|
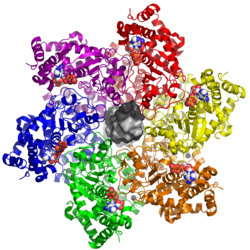
| ||
| nach PDB 5TCT, mit einer zentralen DNA-Doppelhelix (von oben) | ||
| Andere Namen |
Large T antigen | |
| Masse/Länge Primärstruktur | 708 Aminosäuren, 81.582 Da | |
| Sekundär- bis Quartärstruktur | Dodekamer | |
| Bezeichner | ||
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 3.6.4.- | |
Eigenschaften Bearbeiten
Das große T-Antigen dient dem SV 40 zur Regulierung der viralen Replikation. Weiterhin transformiert es die Wirtszelle.[4] Es wird als replikationsrelevantes Protein zu Beginn des viralen Replikationszyklus gebildet (im Gegensatz zu den Kapsidproteinen) und ist essentiell für die virale Replikation.[3]
Das große T-Antigen bringt ruhende Zellen zur Zellteilung durch Überwindung der Checkpoints der Zelle. Dabei bindet es an pRB (synonym RB1) und p53 und bewirkt die Dissoziation des Elongationsfaktors E2F1 von pRB, wodurch die Genexpression von E2F1-regulierten Genen der S-Phase eingeleitet wird. Die Bindung an pRB und p53 erzeugt die transformierende Wirkung.[5] Bei der Erforschung des großen T-Antigens wurde p53 im Jahr 1979 entdeckt.[6][7]
Daneben hemmt das große T-Antigen die Bindung von p53 an DNA, wodurch p53 an der Aktivierung der Genexpression gehemmt wird. Das große T-Antigen wirkt in der Einleitung der Transkription analog zu TAF (TFIID-assoziierter Faktor) für alle drei zellulären RNA-Polymerasen durch Stabilisierung des TBP-TFIIA-Proteinkomplexes an Promotoren. Weiterhin hemmt es die HDAC1-vermittelte Deacetylierung von Histonen, wodurch die Transkription aktiviert wird. Zudem fördert es die Aktivierung des Promotors von Cyclin A über seine J-Proteindomäne. Vermutlich hemmt es die wachstumshemmende Wirkung der E3-Ubiquitinligase Cul7. Die Isoform 17kT des großen T-Antigens führt RBL2 dem Abbau zu und fördert somit das Zellwachstum. Es bindet zweiwertige Zinkionen und ist glykosyliert, acetyliert und phosphoryliert.[8]
Im Genom des SV40 bindet das große T-Antigen an zwei benachbarten Stellen des Replikationsursprungs von SV40 (SV40 ori von engl. origin of replication). Die Verschiebung der Replikationsgabel wird durch eine Helikase-Aktivität des großen T-Antigens vermittelt. Durch die Stabilisierung von TBP und TFIIA aktiviert das große T-Antigen die Transkription der spät gebildeten viralen mRNA.[8]
Neben der Immortalisierung und der Transformation führt das große T-Antigen zu einer gestörten Differenzierung, zum Verlust der DNA-Reparatur und zu einer Instabilität des Karyotyps.[9]
Struktur Bearbeiten
Das große T-Antigen besitzt vier verschiedene konservierte Proteindomänen, verbunden durch unstrukturierte Bereiche: die J-Domäne (ein Chaperon vom Typ DnaJ), die Ori-bindende Domäne (OBD), die Zink-bindende Domäne (für die Bildung von Dodekameren des großen T-Antigens aus zwei hexameren Ringen) und die ATPase vom Typ AAA+. Die Zink-bindende Domäne und die ATPase bilden zusammen die Helikase in der dodekameren Form, welche über die OBD an den Replikationsursprung der viralen DNA bindet.[3] Die ATPase liefert durch die Hydrolyse von Adenosintriphosphat (ATP) die Energie für die Helikase.[3] Einzigartigerweise für ATPasen vom Typ AAA+ trennt sie auch die Doppelstränge der viralen DNA.[3]
Sowohl die unstrukturierten Bereiche als auch die J-Domäne dienen Protein-Protein-Interaktionen mit zellulären Proteinen. Die J-Domäne bindet an HSP70-Vertreter. Die ATPase bindet an die Topoisomerase 1 und p53. Die Ori-bindende Domäne dient Protein-DNA-Interaktionen mit der non-coding control region im Genom des SV40 und bindet an Replication Protein A und Nbs1. Manche große T-Antigene der Polyomaviren besitzen am C-Terminus eine essentielle Sequenz unklarer Funktion, die phosphoryliert wird.[3] Im ersten unstrukturierten Bereich befindet sich eine Signalsequenz für den Transport in den Zellkern.[3]
In manchen Polyomaviren werden die Homologe des großen T-Antigens durch alternatives Spleißen in einer verkürzten Form ohne Zink-bindende Domäne und ohne ATPase-Domäne gebildet, das nur an der Transformation und nicht an der Replikation beteiligt ist.[11][12] Das große T-Antigen besitzt mit dem kleinen T-Antigen die gleichen ersten 80 Aminosäuren.[13] Im murinen Polyomavirus (MPyV) und manchen anderen Polyomaviren (nicht aber SV40) wird zudem noch das mittlere T-Antigen gebildet.[14] Die Mechanismen der Transformation durch T-Antigene von Polyomaviren sind unterschiedlich.[3][15] Das kleine T-Antigen verstärkt die Transformation der Zelle durch das große T-Antigen.
Polyomaviren werden anhand der DNA-Sequenz des großen T-Antigens klassifiziert.[16] Allerdings unterscheidet sich die Phylogenetik, wenn man das Kapsidprotein untersucht.[17]
Weblinks Bearbeiten
Einzelnachweise Bearbeiten
- ↑ Y. Katakura, S. Alam, S. Shirahata: Immortalization by gene transfection. In: Methods in cell biology. Band 57, 1998, S. 69–91, ISSN 0091-679X. PMID 9648100.
- ↑ M. I. Maqsood, M. M. Matin, A. R. Bahrami, M. M. Ghasroldasht: Immortality of cell lines: challenges and advantages of establishment. In: Cell biology international. Band 37, Nummer 10, Oktober 2013, S. 1038–1045, doi:10.1002/cbin.10137, PMID 23723166.
- ↑ a b c d e f g h P. An, M. T. Sáenz Robles, J. M. Pipas: Large T antigens of polyomaviruses: amazing molecular machines. In: Annual review of microbiology. Band 66, 2012, S. 213–236, doi:10.1146/annurev-micro-092611-150154, PMID 22994493.
- ↑ D. Ahuja, M. T. Sáenz-Robles, J. M. Pipas: SV40 large T antigen targets multiple cellular pathways to elicit cellular transformation. In: Oncogene. Band 24, Nummer 52, November 2005, S. 7729–7745, doi:10.1038/sj.onc.1209046, PMID 16299533.
- ↑ J. Cheng, J. A. DeCaprio, M. M. Fluck, B. S. Schaffhausen: Cellular transformation by Simian Virus 40 and Murine Polyoma Virus T antigens. In: Seminars in cancer biology. Band 19, Nummer 4, August 2009, S. 218–228, doi:10.1016/j.semcancer.2009.03.002, PMID 19505649, PMC 2694755 (freier Volltext).
- ↑ D. P. Lane, L. V. Crawford: T antigen is bound to a host protein in SV40-transformed cells. In: Nature. Band 278, Nummer 5701, März 1979, S. 261–263, PMID 218111.
- ↑ D. I. Linzer, A. J. Levine: Characterization of a 54K dalton cellular SV40 tumor antigen present in SV40-transformed cells and uninfected embryonal carcinoma cells. In: Cell. Band 17, Nummer 1, Mai 1979, S. 43–52, PMID 222475.
- ↑ a b Large T antigen - Simian virus 40 (SV40). In: Uniprot.org. 20. Juni 2018, abgerufen am 3. Mai 2018 (englisch).
- ↑ C. D. Toouli, L. I. Huschtscha, A. A. Neumann, J. R. Noble, L. M. Colgin, B. Hukku, R. R. Reddel: Comparison of human mammary epithelial cells immortalized by simian virus 40 T-Antigen or by the telomerase catalytic subunit. In: Oncogene. Band 21, Nummer 1, Januar 2002, S. 128–139, doi:10.1038/sj.onc.1205014, PMID 11791183.
- ↑ W. Lilyestrom, M. G. Klein, R. Zhang, A. Joachimiak, X. S. Chen: Crystal structure of SV40 large T-antigen bound to p53: interplay between a viral oncoprotein and a cellular tumor suppressor. In: Genes & development. Band 20, Nummer 17, September 2006, S. 2373–2382, doi:10.1101/gad.1456306, PMID 16951253, PMC 1560412 (freier Volltext).
- ↑ D. Topalis, G. Andrei, R. Snoeck: The large tumor antigen: a "Swiss Army knife" protein possessing the functions required for the polyomavirus life cycle. In: Antiviral research. Band 97, Nummer 2, Februar 2013, S. 122–136, doi:10.1016/j.antiviral.2012.11.007, PMID 23201316.
- ↑ U. Moens, M. Van Ghelue, M. Johannessen: Oncogenic potentials of the human polyomavirus regulatory proteins. In: Cellular and molecular life sciences : CMLS. Band 64, Nummer 13, Juli 2007, S. 1656–1678, doi:10.1007/s00018-007-7020-3, PMID 17483871.
- ↑ M. Van Ghelue, M. T. Khan, B. Ehlers, U. Moens: Genome analysis of the new human polyomaviruses. In: Reviews in medical virology. Band 22, Nummer 6, November 2012, S. 354–377, doi:10.1002/rmv.1711, PMID 22461085.
- ↑ M. M. Fluck, B. S. Schaffhausen: Lessons in signaling and tumorigenesis from polyomavirus middle T antigen. In: Microbiology and molecular biology reviews : MMBR. Band 73, Nummer 3, September 2009, S. 542–63, Table of Contents, doi:10.1128/MMBR.00009-09, PMID 19721090, PMC 2738132 (freier Volltext).
- ↑ G. Stakaitytė, J. J. Wood, L. M. Knight, H. Abdul-Sada, N. S. Adzahar, N. Nwogu, A. Macdonald, A. Whitehouse: Merkel cell polyomavirus: molecular insights into the most recently discovered human tumour virus. In: Cancers. Band 6, Nummer 3, Juni 2014, S. 1267–1297, doi:10.3390/cancers6031267, PMID 24978434, PMC 4190541 (freier Volltext).
- ↑ Study Group of the International Committee on Taxonomy of Viruses, S. Calvignac-Spencer, M. C. Feltkamp, M. D. Daugherty, U. Moens, T. Ramqvist, R. Johne, B. Ehlers: A taxonomy update for the family Polyomaviridae. In: Archives of virology. Band 161, Nummer 6, Juni 2016, S. 1739–1750, doi:10.1007/s00705-016-2794-y, PMID 26923930.
- ↑ C. B. Buck, K. Van Doorslaer, A. Peretti, E. M. Geoghegan, M. J. Tisza, P. An, J. P. Katz, J. M. Pipas, A. A. McBride, A. C. Camus, A. J. McDermott, J. A. Dill, E. Delwart, T. F. Ng, K. Farkas, C. Austin, S. Kraberger, W. Davison, D. V. Pastrana, A. Varsani: The Ancient Evolutionary History of Polyomaviruses. In: PLoS pathogens. Band 12, Nummer 4, April 2016, S. e1005574, doi:10.1371/journal.ppat.1005574, PMID 27093155, PMC 4836724 (freier Volltext).